
폐암 환자에서 암 돌연변이를 분석해 EGFR 계열 항암제에 대한 내성을 쉽고 정확하게 파악할 수 있는 기술이 개발됐다.
연세대 의대는 약리학교실 김형범 교수, 오형철 강사, 외과학교실 이승호 강사, 김영광 가톨릭대의대 병리학교실 교수팀이 차세대 유전자교정 기술 '프라임 편집기'를 적용해 폐암 환자의 돌연변이 유발·검출 기술을 개발하고 이를 통해 약물에 대한 내성 수준까지 확인하는 데에 성공했다고 22일 발표했다. 이번 연구 결과는 국제학술지 '네이처 바이오테크놀로지' 최신호에 실렸다.
폐암 환자의 30% 정도에게서 확인되는 표피생장인자수용체(EGFR) 변이는 ‘티로신키나제 억제제(TKI) 등 표적치료제 사용 여부를 결정하는 중요한 기준이다. 다만 표적치료제는 치료 초기 효과가 좋다가 1~2년 뒤 다른 돌연변이가 생기는 내성 탓에 효과가 떨어지는 게 문제다.
지금은 약물 내성과 돌연변이 간 관계를 규명하기 위해 약제에 노출된 환자 종양 조직을 채취한 뒤 저항성 유전 변이를 발굴했다. 환자마다 보유한 돌연변이가 다르고 사례를 충분히 확보하는 것도 힘들어 단일 돌연변이로만 내성을 평가하는 데엔 한계가 있었다.
연구팀은 프라임 편집기와 인공지능 기술을 활용해 새로운 돌연변이 검출 기술 'PEER-seq'을 개발했다.
모든 형태의 단일염기서열 변이(SNV)를 활용한 것은 물론 유전자 변이가 생겨도 단백질 아미노산 배열은 바뀌지 않는 동의돌연변이 정보도 반영했다. 이를 통해 PEER-seq 기술로 모든 종양 변이 중 95% 이상을 검출할 수 있다는 것을 입증했다. 해당 돌연변이가 항암제 내성에 미치는 영향 정도도 평가할 수 있었다.
유전자가위로 세포 수 변화를 간접 추적한 기존 방식과 달리 유전체 속 직접 돌연변이의 약제 반응 정도를 확인할 수 있어 높은 정확도로 기능을 평가할 수 있다고 연구진은 설명했다.
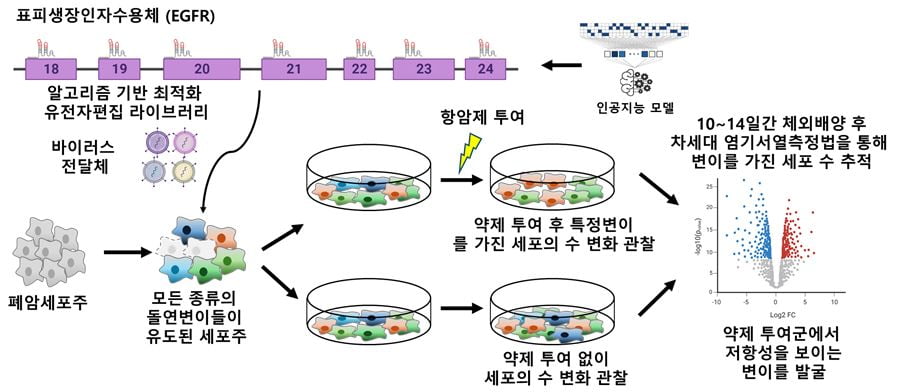
연구팀은 이렇게 구축한 기술을 활용해 돌연변이가 나온 세포에 폐암 EGFR 표적치료제인 타그리소(성분명 오시머티닙)와 리보세라닙(성분명 아파티닙)을 투여한 뒤 2476개 변이가 항암제에 저항성을 보이는지 평가했다.
EGFR 유전자에 가장 많이 발생하는 T790M 변이가 동반된 상태에서 다른 돌연변이가 유도된 ‘복합 변이’ 조합이 항암제 내성에 어떤 영향을 미치는 지도 분석했다. 이를 통해 T790M이 동반 여부에 따라 복합 변이 조합의 항암제 내성도 차이난다는 것을 확인했다.
연구팀에 따르면 2476개 변이와 3가지 약물 투여 조합에서 그동안 항암제 저항성 여부가 밝혀지지 않았던 46개 조합의 저항성 여부를 새롭게 확인했다.
기존 진단키트로는 찾지 못했던 새로운 약제 저항성 변이(K754Q, G930R, E931K)도 찾아냈다. 연구팀은 이번 연구를 통해 확인된 변이들의 특징이 기존 보고 내용과도 일치한다는 것을 확인했다.
김형범 교수는 "PEER-seq 기술로 많은 돌연변이의 약물 저항성을 높은 정확도로 한번에 분석할 수 있게 됐다"며 "폐암 치료뿐 아니라 다른 암종에서도 다양한 항암제와 변이 조합을 평가하는데 활용할 수 있다"고 했다. 신약개발, 환자 맞춤형 치료 플랫폼 구축 등 정밀의학 실현에 폭넓게 활용될 것으로 내다봤다.
이지현 기자 bluesky@hankyung.com

 10 hours ago
3
10 hours ago
3















 English (US) ·
English (US) ·